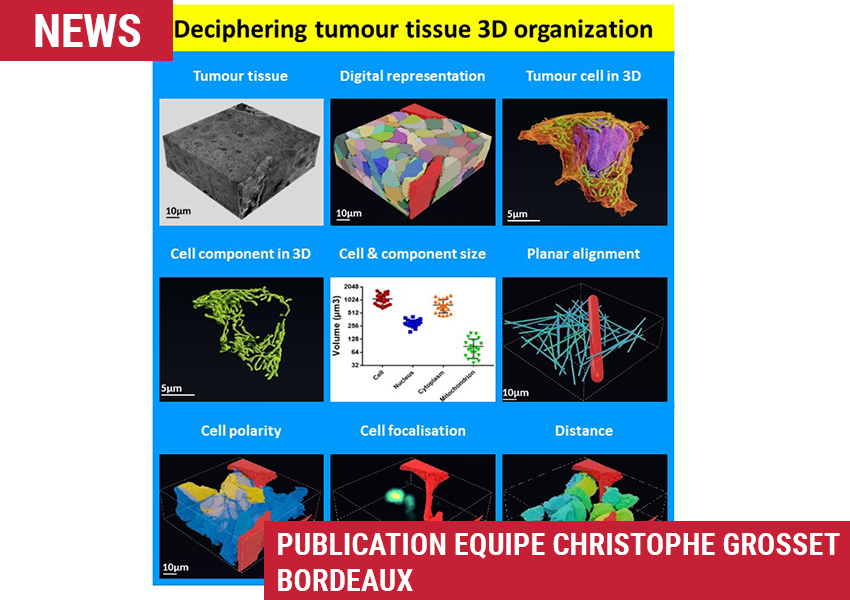
Le 13 décembre 2021, le consortium pluridisciplinaire coordonné par Christophe Grosset (Equipe MIRCADE, BRIC – Inserm U1312, ex- BMGIC – Inserm U1035, à Bordeaux) a publié un article original intitulé « Deciphering tumour tissue organization by 3D electron microscopy and machine learning » dans le journal Communications Biology.
Depuis 2015, l’équipe MIRCADE (Méthodes et Innovations pour la Recherche sur les CAncers De l’Enfant) étudie la biologie de plusieurs tumeurs solides chez l’enfant, dont l’hépatoblastome, un cancer primaire du foie. A la suite d’observations faites en laboratoire, Christophe Grosset s’est interrogé sur les paramètres contrôlant l’organisation interne d’un tissu tumoral, sachant qu’ils sont très mal connus.
Pour répondre à cette question, il s’est entouré de chercheurs et ingénieurs des Départements Sciences Biologiques et Médicales, Bordeaux Neurocampus et Sciences de l’ingénierie et du Numérique de l’Université de Bordeaux, parmi lesquels Baudouin Denis de Senneville (Institut de Mathématiques), Fatma Zohra Khoubai (Equipe MIRCADE), Jean Ripoche (Inserm, Université de Bordeaux), Marc Bevilacqua et Etienne Gontier (Bordeaux Imaging Center, Pôle Microscopie Electronique). Il a également sollicité les compétences d’Alexandre Labedade, Infographiste indépendant, de Christophe Chardot et Sophie Branchereau, tous deux chirurgiens pédiatriques à l’AP-HP, ainsi que de Kathleen Flosseau et Stefano Cairo de la société XenTech. Après analyses des échantillons biologiques par la technique « Serial block-face scanning electron microscopy » au BIC, le consortium a développé une méthodologie basée sur la segmentation semi-automatique des principaux éléments structuraux d’un tissu tumoral tels que les capillaires sanguins, les cellules, les noyaux et les mitochondries (https://github.com/bsenneville/Onconanotomy).
Comme la segmentation manuelle est trop laborieuse pour segmenter des milliers d’éléments, le consortium a entrainé un réseau neuronal artificiel de type U-net à la segmentation automatique des éléments du tissu, générant ainsi plusieurs banques de données d’apprentissage (protégés par brevet, https://github.com/bsenneville/Onconanotomy). Par cette approche, 182 cellules, 113 noyaux et 3 portions d’un capillaire sanguin ont été segmentés, puis analysés par des approches mathématiques. Le consortium a alors découvert (i) que les capillaires sanguins et des structures proches des canalicules biliaires influencent l’alignement planaire, la polarité et la taille des cellules tumorales et de leur réseau mitochondrial, et (ii) que la taille des cellules d’hépatoblastome est corrélée à celle de leur noyau, de leur cytoplasme et de leur réseau mitochondrial. Par ce travail, le consortium apporte quelques infirmations sur les paramètres bioarchitecturaux contrôlant l’organisation 3D d’un tissu tumoral, ouvrant ainsi la voie à de futures investigations dans un domaine émergent appelé « Onconanotomy ».

Article réalisé avec le Département Sciences Biologiques et Médicales de l’Université de Bordeaux